PDF-Version
Neues aus der Forschung
Die Evolution des fehlangepassten Enzyms RuBisCO: Zurück in die Zukunft der Photosynthese
Die Rekonstruktion uralter Enzyme enthüllt, wie sich die Photosynthese an das Auftreten von Sauerstoff anpassen konnte
Irdisches Leben kommt nicht ohne Photosynthese betreibende Organismen wie Pflanzen und Algen aus, die Kohlendioxid einfangen und in energiereiche Assimilate (Stärke) umwandeln. Das Herzstück dieses Prozesses ist ein Enzym namens RuBisCO1), das jährlich 400 Milliarden Tonnen Kohlendioxid bindet und in Biomasse umsetzt. Evolutionär bedingt ist das Enzym jedoch mit einem schweren Mangel behaftet, die Photosynthese bleibt daher notorisch ineffizient.
Wie entwickelte sich RuBisCO, und wie passte es sich den Veränderungen im Laufe der Erdgeschichte an? In Zusammenarbeit mit Prof. Tobias ERB am MPI Marburg und der Universität Singapur ist es unserer Forschungsgruppe gelungen, Milliarden Jahre alte Enzyme zu rekonstruieren, im Labor nachzubauen und zu untersuchen (SCHULZ et al. 2022). Unsere Ergebnisse liefern Erkenntnisse zur Evolution der modernen Photosynthese sowie neue Ansätze für ihre Optimierung.
Urzeitliche Fehlanpassung des Enzyms RuBisCO
Der Prozess der Kohlenstoff-Fixierung ernährt sprichwörtlich alles Leben auf der Erde. Der verantwortliche Stoffwechselweg ist der sogenannte Calvin-Benson-Zyklus, auch Dunkelreaktion genannt. Herzstück des Prozesses ist das Enzym RuBisCO, welches CO2 bindet und es in Form einer Carboxylgruppe auf einen aktivierten C5-Zucker überträgt. Dieses Produkt ist instabil und zerfällt rasch in zwei Moleküle eines C3-Körpers. In einer verzweigten Reaktionsfolge wird daraus über verschiedene Zwischenstufen wieder der ursprüngliche, aktivierte C5-Zucker erzeugt. Netto fällt bei dieser Reaktion ein bestimmter C3-Zucker an (GAP), der zur Herstellung von Assimilaten (zunächst Glucose, danach Stärke) genutzt wird.
RuBisCO ist uralt und entstand im Ur-Stoffwechsel vor drei bis vier Milliarden Jahren, noch bevor es auf der Erde nennenswerte Mengen an Sauerstoff gab. Die Evolution passte RuBisCO somit an die Bedingungen der frühen Erde an. Doch mit der Entwicklung der sauerstoffproduzierenden Photosynthese und dem damit verbundenen Anstieg des Sauerstoffs in der Atmosphäre begann RuBisCO unversehens damit, eine Fehlreaktion zu katalysieren: Das Enzym verwechselt nämlich O2 mit CO2 und begann plötzlich damit, Stoffwechselprodukte zu erzeugen, die für die Zelle giftig sind (Abb. 2). Damit die Pflanzen überleben konnten, mussten sie einen Stoffwechselweg etablieren, der die toxischen Nebenprodukte mit großem Energieaufwand entgiftet.
In dieser sogenannten Photorespiration geht ein Drittel der durch die Lichtreaktion eingefangenen Gesamtenergie wieder verloren. In dieser "Lichtatmung" wird wieder Kohlendioxid freigesetzt und Sauerstoff verbraucht. Des Weiteren muss RuBisCO extrem langsam arbeiten, um die Fehlerrate niedrig zu halten; es setzt nur wenige Kohlendioxidmoleküle pro Sekunde um. Diese Substrat-Ungenauigkeit beeinträchtigt RuBisCO bis heute, und sie vermindert die Effizienz der Photosynthese enorm: Bereits eine Halbierung der vorindustriellen CO2-Konzentration würde ausreichen, um die Reaktion zum Erliegen zu bringen. Der Photosynthese-Motor ruckelt und stolpert.
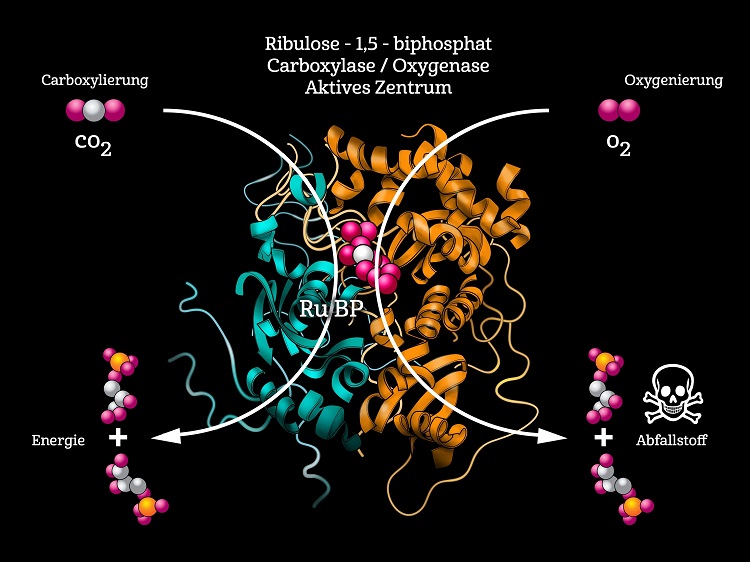
Abb. 2 Die zentrale dimere Einheit aller RuBisCO-Varianten. Sie katalysiert sowohl die nützliche Carboxylierung als auch die schädliche Oxygenierung. Nach TABITA et al. (2008), verändert.
Um der evolutiven Fehlanpassung entgegen zu wirken, müssen die Pflanzen enorme Mengen an RuBisCO in ihren Blättern synthetisieren. Einige Arten schafften es, Mechanismen zu entwickeln, um RuBisCO behelfsweise gegen Sauerstoff abzuschirmen. Dazu wird das CO2 an einen C4-Zucker gebunden und in die Bündelscheidenzellen transportiert, wo RuBisCO aktiv ist. Dort gibt der Zucker das CO2 wieder ab, wodurch die CO2-Konzentration am Wirkungsort von RuBisCO ansteigt. Dadurch kommt das Enzym mit weniger Sauerstoff in Kontakt. Pflanzen, die diesen verbesserten Photosynthese-Motor benutzen, nennt man C4-Pflanzen.2) Sie machen allerdings nur 10% aller Landpflanzen aus. Nutzpflanzen wie Weizen, Roggen, Gerste, Hafer, Kartoffel, Reis sowie weltweit alle Baumarten gehören nicht dazu.
Die Wiedererweckung uralter Proteine im Labor
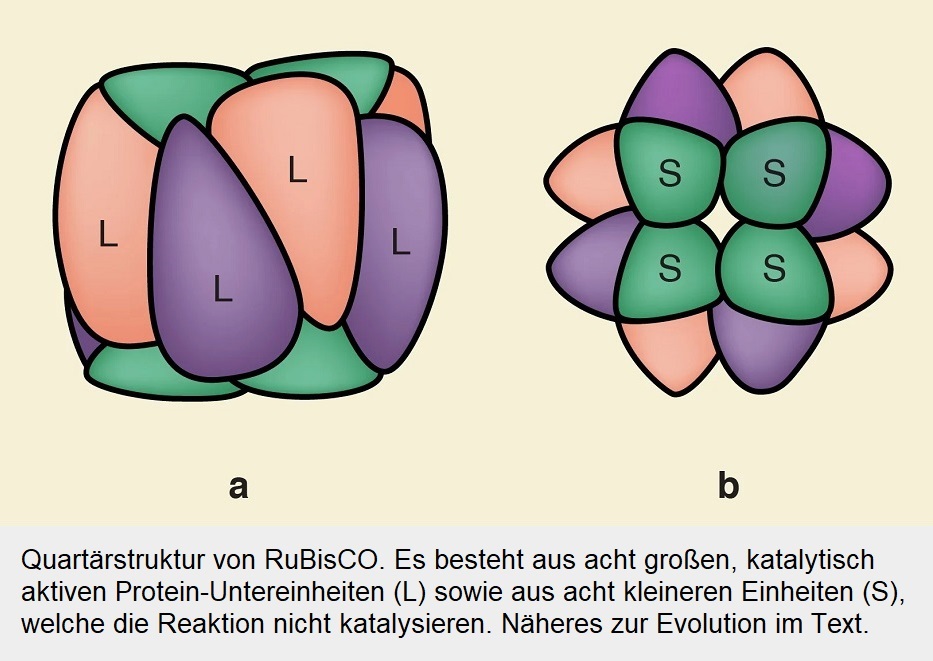
Obwohl die RuBisCO-Varianten von Organismen in sauerstoffhaltigen Umgebungen im Laufe der Zeit immer spezifischer für CO2 wurden, konnte keines von ihnen die hinderliche Sauerstoff-Bindereaktion ganz abschaffen. Die molekularen Ursachen für die Verbesserung der CO2-Spezifität sind noch weitgehend unbekannt. Dabei sind sie von großem Interesse, wenn man darauf abzielt, die Effizienz der Photosynthese zu verbessern. Es ist auffällig, dass RuBisCO-Formen, die eine erhöhte CO2-Spezifität aufweisen, eine neue Proteinkomponente mit bislang unbekannter Funktion aufweisen. Es handelt sich um die kleinen Untereinheiten (S), die in der eingangs abgebildeten Schemazeichnung grau dargestellt sind. Man vermutete, dass diese Komponente an der Steigerung der Spezifität beteiligt ist. Doch das war schwer zu ermitteln, da ihre Entstehung Milliarden Jahre zurückliegt.
Um dieses entscheidende Ereignis in der Evolution spezifischerer RuBisCO-Varianten besser zu verstehen, griff unsere Forschungsgruppe am MPI in Kooperation mit der Nanyang Technological University in Singapur auf einen statistischen Algorithmus zurück. Mit ihm rekonstruierten wir am Computer diejenigen Formen der RuBisCOs, die vor Milliarden von Jahren existierten, bevor der Sauerstoffgehalt anstieg. Im Team mit Prof. Dr. Tobias ERB und mir gelang es, die uralten Proteine im Labor nachzubauen und ihre Eigenschaften zu untersuchen.
Uns interessierte insbesondere, ob und wie die neue Strukturkomponente mit der Evolution der höheren Spezifität in Zusammenhang steht. Die Antwort war überraschend: Wir hatten erwartet, dass die neue Komponente den Sauerstoff irgendwie direkt aus dem katalytischen Zentrum von RuBisCO ausschließt. Das tut sie aber nicht. Stattdessen scheint die neue Untereinheit als Modulator für die Evolution zu fungieren: Sie veränderte den Effekt nachfolgender Mutationen auf die katalytische Untereinheit von RuBisCO, so dass zuvor unbedeutende Mutationen auf einmal einen großen Einfluss auf die Spezifität hatten. Es scheint also, dass das Vorhandensein der neuen Untereinheit das evolutionäre Potenzial von RuBisCO veränderte.
Nicht reduzierbar komplex: Ein Enzym wird abhängig von seiner neuen Untereinheit
Diese Funktion als "evolutionärer Modulator" erklärt auch ein anderes Rätsel dieser Proteinkomponente: Alle modernen RuBisCOs, die diese eingebaut haben, sind in ihrer Funktion vollständig von ihr abhängig, obwohl einfachere Formen von RuBisCO auch ohne sie funktionieren. Sie sind also "nicht reduzierbar komplex". Der Modulationseffekt liefert den Grund: Als RuBisCO an diese kleine Proteinkomponente gebunden war, konnte das Enzym Mutationen tolerieren, die alleine katastrophal gewesen wären. Sobald sich genügend solcher Mutationen angesammelt hatten, wurde RuBisCO von der neuen Untereinheit abhängig.
Unsere Ergebnisse, die in der jüngsten Ausgabe von "Science" veröffentlicht wurden, geben damit Aufschluss über die bisher ungelöste Frage, warum RuBisCO die neue Proteinkomponente seither beibehielt: Die Tatsache, dass dieser Zusammenhang bis jetzt nicht verstanden war, unterstreicht die Bedeutung der evolutionären Analyse für das Verständnis der Biochemie um uns herum.
Die Entstehungsgeschichte von Biomolekülen wie RuBisCO kann uns so viel darüber lehren, warum sie so sind, wie sie heute sind. Und es gibt immer noch so viele biochemische Phänomene, deren Entwicklungsgeschichte uns völlig unbekannt ist. Es ist also eine sehr aufregende Zeit für einen Evolutionsbiochemiker: Fast die gesamte molekulare Geschichte der Zelle wartet noch darauf, aufgeklärt zu werden.
RuBisCO: Evolution vom einfachen Enzym zum Proteinkomplex
Wie wurde RuBisCO überhaupt zum wichtigsten CO2-fixierenden Biokatalysator in der Natur, und wie konnte es eine derart hohe Komplexität erreichen? Aufschluss darüber geben RuBisCO-ähnliche Proteine (RLP), die zusammen mit den heute bekannten RuBisCO-Varianten (Form I, II und III) die RLP/RuBisCO-Superfamilie bilden. Während jedoch die RuBisCO-Formen I, II und III CO2-Fixierungsreaktionen katalysieren, unterhält das RLP (auch: RuBisCO-Form IV) grüner Schwefelbakterien Reaktionen des Schwefelstoffwechsels.
Die RuBisCOs der Form III, die in Archaeen gefunden wurden, stellen wahrscheinlich die älteste Form des Enzyms dar. Es ist nicht bekannt, dass RuBisCOs der Form III im Calvin-Zyklus zur autotrophen CO2-Fixierung wirken; vielmehr dienen sie der Assimilation von Ribonukleosiden. Im sogenannten Pentosebisphosphat-Weg in Archaeen, denen der "kanonische" Pentosephosphat-Weg fehlt, wird die Riboseeinheit von Adenosin, Guanosin und Uridin durch RuBisCO metabolisiert.
Ein weiterer interessanter Punkt in diesem Zusammenhang ist die Tatsache, dass der Calvin-Zyklus auch heute nicht nur bei der autotrophen CO2-Fixierung funktioniert. Während des anaeroben Wachstums von α-Proteobakterien wird der Calvin-Zyklus hauptsächlich zum Recycling von Redox-Cofaktoren auf Wachstumssubstraten verwendet, die stärker reduziert sind als der durchschnittliche Zellkohlenstoff. Darüber hinaus beherbergen mehrere nicht-autotrophe Bakterien im Boden RuBisCO-Gene, die auch dem Redoxausgleich und der Kohlenstofffixierung dienen.
Alle heutigen RuBisCOs (einschließlich der RLPs) haben ein gemeinsames Merkmal: Ihre aktiven Zentren werden an der Grenzfläche zwischen zwei großen Untereinheiten gebildet, die alle für die Katalyse erforderlichen Reste beisteuern. Jenseits dieses homodimeren Enzymkerns (L2) wurden RuBisCOs jedoch während der Evolution in höheren oligomeren Zuständen organisiert. Es ist bekannt, dass RuBisCOs der Formen II und III mit höherer (L2)n-Stöchiometrie (mit n bis zu 5) auftreten, und RuBisCOs der Form I sind in vier L2-Dimeren organisieren, die sich mit einer zusätzlichen kleinen Untereinheit (S) zu einem L8S8-RubisCO-Komplex zusammenlagern (Abb. 3).
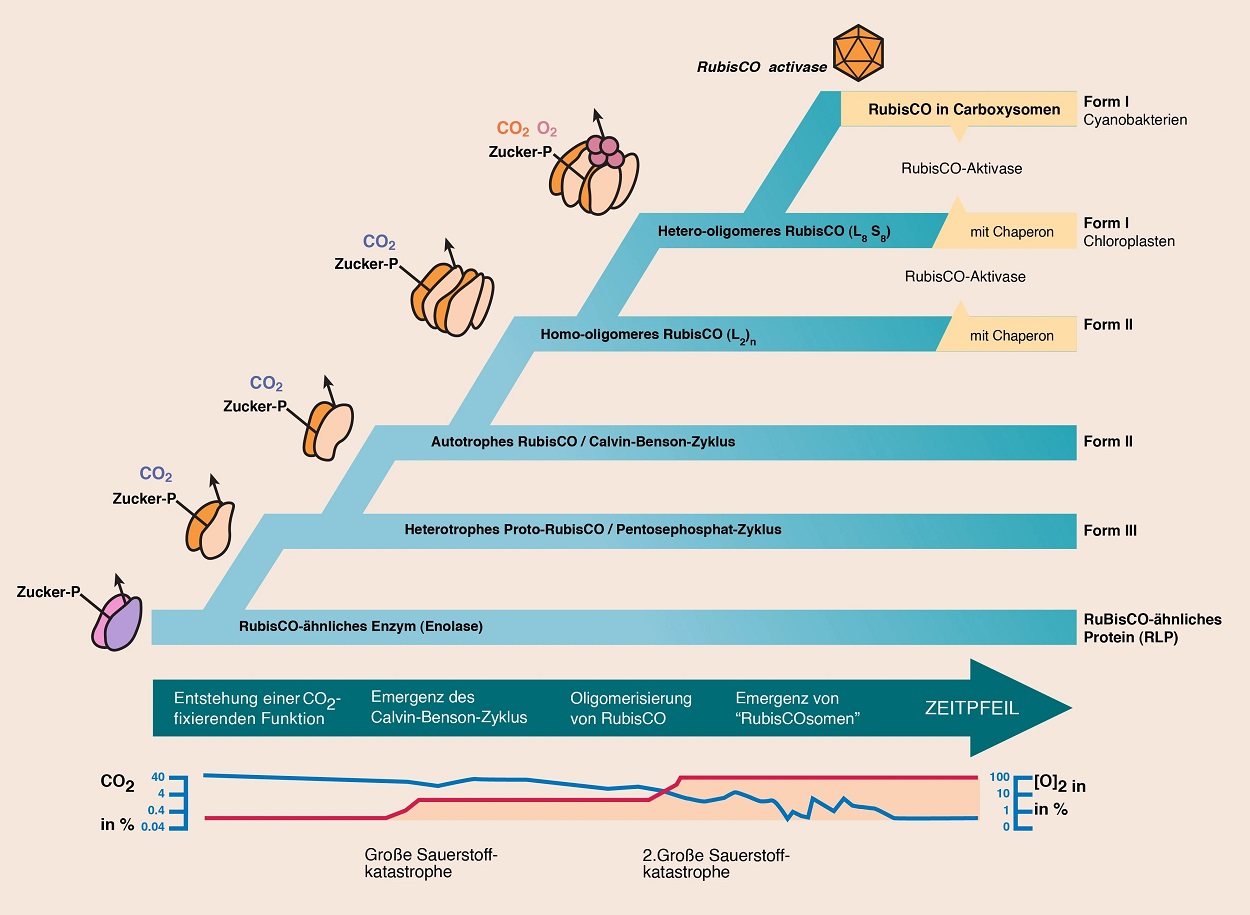
Abb. 3 Modell zur Evolution von RuBisCO, ausgehend vom einem nicht-CO2-fixierenden RLP-Protein mit Enolase-Aktivität bis zum modernen RuBisCO (Form I), das in Chloroplasten und cyanobakteriellen Carboxysomen anzutreffen ist. Verändert nach ERB & ZARZYCKI (2018, S. 103).
Wie oben angedeutet, erhöht das Vorhandensein der zusätzlichen kleinen Untereinheiten die katalytische Effizienz und Spezifität. Insbesondere durch das Aufkommen der oxygenen Photosynthese und der großen Sauerstoffkatastrophe wurde RuBisCO einem starken evolutiven Druck ausgesetzt, seine CO2-Fixierungseffizienz in Gegenwart von Sauerstoff aufrechtzuerhalten.
RuBisCO erlangte einen evolutionären Zustand, in dem weder die Aktivität noch die Spezifität des Enzyms weiter verbessert werden konnte, ohne den jeweils anderen Parameter negativ zu beeinflussen. Das erklärt, warum es der Evolution nicht gelang, die hinderliche Sauerstoff-Bindereaktion nachträglich wieder abzuschaffen. Es blieb nur die Möglichkeit neue Strategien zu entwickeln, um das katalytische Dilemma zu überwinden und die Enzymleistung weiter zu verbessern. Die evolutionären Anpassungen gipfelten in der Bildung eines facettenreichen RuBisCO-Komplexes.
Zusammenfassend legen die erwähnten Fakten sowie eine phylogenetische Auswertung der Gene, die für diese Proteine codieren, eine Evolution in sechs Hauptschritten nahe (vgl. Abb. 3; ERB & ZARZYCKI 2018, S.103):
Schritt 1: Die Proteine der RLP/RuBisCO-Superfamilie stammen von einem nicht- CO2-bindenden Enzym vom Enolase-Typ ab. Innerhalb dieser Familie entstand zunächst eine Form mit Carboxylase-Aktivität (RuBisCO-Form IV).
Schritt 2: Bei diversen Archaeen ("Archae-Bakterien") entwickelte sich die RuBisCO-Form III als CO2-fixierendes Enzym in einem alten, wahrscheinlich archaealen Nukleotid-Assimilationsweg (heterotrophes Proto-RuBisCO; RuBisCO-Form III).
Schritt 3: Es entwickelte sich die autotrophe Funktion (RuBisCO-Form II).
Schritte 4 bis 6: Die hetero-oligomeren RuBisCO-Formen I entwickelten sich durch das Entstehen von Homo-oligomerisierung und die Hinzuziehung einer weiteren, katalytisch nicht aktiven Untereinheit. Diese Veränderungen erhöhten die CO2-Spezifizität schon vor der großen Sauerstoffkatastrophe und ermöglichten dadurch die spätere Verbreitung der oxygenen Photosynthese.
ECR-Enzyme: intelligente Alternativen zu RuBisCO
Mit Blick auf die energetisch und katalytisch ungünstige Fehlreaktion mit Sauerstoff hat sich mein Kollege Prof. Tobias ERB auf die Erforschung von Alternativen zur RuBisCO verlegt. Denn die gibt es durchaus: Enzyme aus der Klasse der Enoyl-CoA Carboxylasen/Reduktasen (ECR) arbeiten um mehrere Größenordnungen schneller als die RuBisCO und machen keinen Fehler mit Sauerstoff.
Um herauszufinden, wie sie das bewerkstelligen, untersuchten Kollegen aus ERBs Forschungsgruppe sowie aus Chile und den USA die ECR aus dem Bakterium Kitasatospora setae, die derzeit schnellste bekannte Carboxylase. In einem kombinierten Ansatz aus Strukturbiologie, Biochemie und Computersimulationen vollzogen sie zum ersten Mal nach, wie das Enzym CO2 bindet und umwandelt.
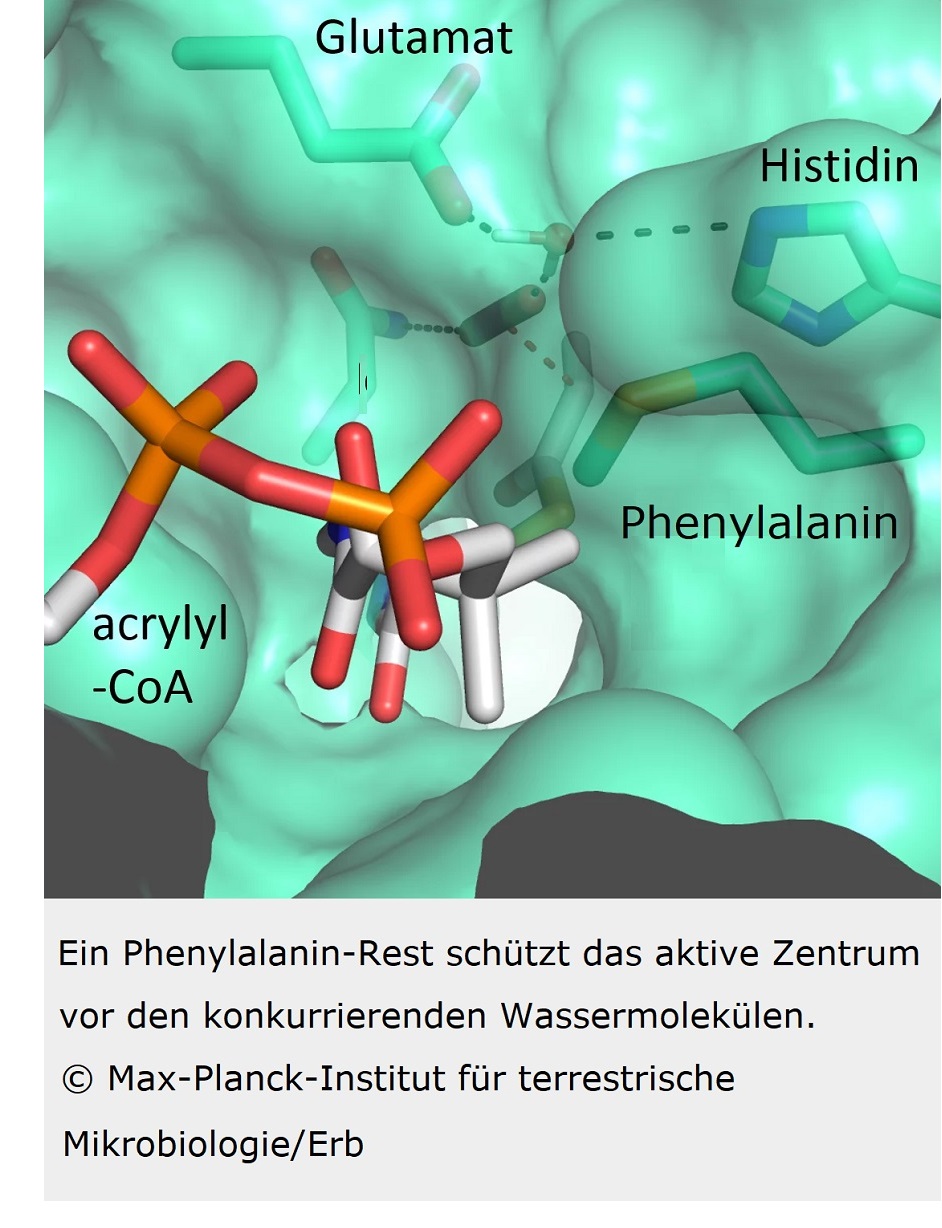
Unsere Kollegen waren erstaunt zu sehen, dass vier Aminosäuren bereits ausreichen, um dem Protein höchstmögliche Kontrolle über das CO2-Molekül zu verleihen (STOFFEL et al. 2019). Drei Aminosäuren – Asparagin, Glutamat und Histidin – verankern das CO2 von zwei Seiten. Eine weitere Aminosäure, ein Phenylalanin, schirmt das gebundene CO2 wie ein Schutzschild gegen Wasser ab, das die Reaktion inhibieren würde (Abb. 4).
Alles in allem ist es gelungen, einen Prozess im Reagenzglas zu bauen, der CO2 besser assimiliert als die in der Natur vorkommenden Wege der Photosynthese. Höchste Robustheit und Energieeffizienz sind die Eigenschaften, die es der künstlichen Photosynthese zu verleihen gilt. Diese Fähigkeit, spezifisch CO2 zu binden, möchten unsere Kollegen auch auf andere Enzyme übertragen. Damit hätten sie für die Optimierung der künstlichen Photosynthese einen viel größeren Spielraum.
Enzym-Engineering und "rationales Design" von RuBisCO
Unsere Studie wiederum liefert wichtige Hinweise darauf, wie die durch RuBisCO katalysierte Photosynthese synthetisch-biologisch verbessert werden könnte: Die Ergebnisse unserer Forschung haben gezeigt, dass die bisherigen Versuche, RuBisCO zu verbessern, möglicherweise an der falschen Stelle ansetzten. Jahrelang konzentrierte sich die Forschung ausschließlich auf die Veränderung von Aminosäuren in RuBisCO selbst. Unsere Arbeit deutet nun darauf hin, dass das Hinzufügen völlig neuer Proteinkomponenten wesentlich zielführender sein könnte und möglicherweise neue Wege in der Evolution eröffnet. Dies ist Neuland für das Enzym-Engineering wie es von Prof. ERB betrieben wird, und wir sind gespannt, wohin es uns führen kann.
Literatur
ERB, T.J. & ZARZYCKI, J. (2018) A short history of RubisCO: the rise and fall (?) of Nature’s predominant CO2 fixing enzyme. Current Opinion in Biotechnology 49, S. 100–107.
GOWIK, U. & WESTHOFF, P. (2011) The path from C3 to C4 photosynthesis. Plant Physiology 155, 56–63.
SAGE, R.F.; SAGE, T.L. & KOCACINAR, F. (2012) Photorespiration and the evolution of C4 photosynthesis. Annual Review of Plant Biology 63, 19–47.
SCHULZ, L.; GUO, Z.; ZARZYCKI, J.; STEINCHEN, W.; SCHULLER, J.M.; HEIMERL, T.; PRINZ, S.; MUELLER-CAJAR, O.; ERB, T.J. & HOCHBERG, G.K.A. (2022) Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science 378, 155–160.
STOFFEL, G.M.M.; SAEZ, D.A., DEMIRCI, H.; VÖGELI, B.; RAO, Y.; ZARZYCKI, J. YOSHIKUNI, Y.; WAKATSUKI, S.; VÖHRINGER-MARTINEZ, E. & ERB, T.J. (2019) Four amino acids define the CO2 binding pocket of enoyl-CoA carboxylases/reductases. PNAS 116, 13964-13969.
TABITA, F.R.; SATAGOPAN, S.; HANSON, T.E.; KREEL, N.E. & SCOTT, S.S. (2008) Distinct form I, II, III, and IV Rubisco proteins from the three kingdoms of life provide clues about Rubisco evolution and structure/function relationships. Journal of Experimental Botany 59, 1515–1524.
UPADHYAY, P.; AGRAWAL, N.; KUMAR, P.Y. & PATEL, R. (2020) The evolutionary path from C3 to C4 photosynthesis: a review. International Journal of Current Microbiology and Applied Sciences 9, 748–762.
Links zum MPI für Terrestrische Mikrobiologie
- Deutsches Press Release: "Zurück in die Zukunft der Photosynthese". https://www.mpi-marburg.mpg.de/1236034/2022-10-a
- Englische Version: https://www.mpg.de/19348003/1010-terr-back-to-the-future-of-photosynthesis-153410-x
- Interview: "Wir können versuchen, die Evolution in eine andere Richtung zu lenken". https://www.mpg.de/19365851/interview-erb
- Research Area Dr. Georg HOCHBERG, Evolutionäre Biochemie: https://www.mpi-marburg.mpg.de/hochberg
Fußnoten
[1] RuBisCO ist die Abkürzung für Ribulose-1,5-bisphosphat-carboxylase/-oxygenase.
[2] Zur Evolution des C4-Stoffwechsels, der sich konvergent in mehreren Zwischenschritten entwickelte, vgl. GOWICK & WESTHOFF (2011), SAGE et al. (2012), UPADHYAY et al. (2020).
Autor: Dr. Georg Hochberg